Chp 06 Alternative Splicing
Molecular Biology of RNA 2nd edition (David Elliott & Michael Ladomery)
Gene number paradox

유전자의 copy number와 complexity 사이에 불일치가 발견된다. 유전자가 더 많다고 무조건 복잡한 것도 그 반대도 아니었던 것이다. 이 현상을 과학은 어떻게 설명할 수 있었을까? 바로 spicing이다. 고등생물일수록(난 아직도 그놈의 고등하다는 말이 뭔지 모르겠지만) non coding gene을 이용하여 적은 수의 유전자로도 복잡한 조절을 할 수 있다고 한다. 바로 Alternative splicing과 같은 방법을 이용해서 말이다!
Alternative Splicing
바로 여기 Cannonical Splicing과 다르게 alternative splicing은 모든 exon이 포함되는 대신 선택적으로 포함되거나 포함되지 않는 것을 의미한다.
 |
모든 exon이 포함된다. |
Alternative splicing의 종류는 다음과 같다. 7공주다.
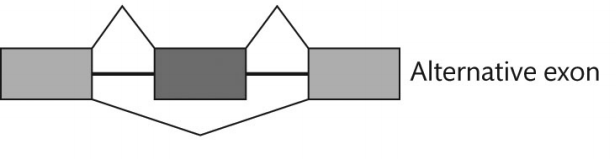 |
exon이 선택적으로 포함된다. |
 |
하나의 exon에 다른 두 개의 5' Splice site가 존재해 선택적으로 splice 된다. |
 |
하나의 exon에 다른 두 개의 3' Splice site가 존재해 선택적으로 splice 된다. |
 |
상호배타적으로 하나의 exon이 선택되면 다른 exon은 선택되지 않는다. |
 |
intron이 제거되지 않고 남는다. intron definition을 이용하는 생명체에서 많이 일어난다. |
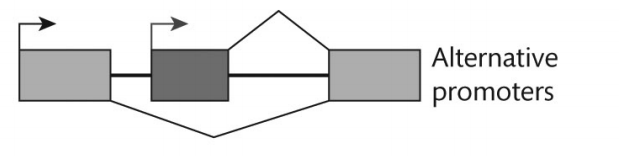 |
spliceosome이 포함되지 않는 과정이다. 엄연히 따지면 promoter는 intron이기에 그림이 잘못되었다고 할 수 있다. |
 |
spliceosome이 포함되지 않는 과정이다. 엄연히 따지면 poly adenylation site는 intron이기에 그림이 잘못되었다고 할 수 있다. |
Alternative splicing이 일어나 형성된 서로 다른 isoform을 detection하기 위해 RNA-seq 등을 수행할 수 있다.

이때 Depth는 DNA에 얼마나 allign 되는지를 설명하고(그 부위가 4번 겹치면 4X...), Breadth는 전체 DNA의 몇 %나 커버하는지를 알려준다. (100% 일치하면 100%, 20% 비면 80%...)
Exon / Intron Definition

사실 완전히 이해하지 못한 개념이라서 틀릴까봐 포스팅을 고민했는데, Splicing에서 꽤나 중심이 되는 개념인 것 같아서 이해한 것을 토대로 포스팅한다.
Splicing 초기에 Exon을 인식하면 Exon-definition을 이용한 Splicing, intron을 인식하면 intron-definition을 이용한 splicing이라고 한다.
eukaryote은 짧은 exon과 상대적으로 긴 intron을 가지고 있기 때문에 intron을 인식하는 일은 무척 어려운 일이다. 따라서 exon을 먼저 인식해서 mark해둔 다음 그것을 기준으로 intron을 인식해 intron을 제거하게 된다. (Exon definition) 이러한 Exon definition을 이용한 splicing에서는 Exon skipping이 가장 많이 일어나는 에러가 된다. (Exon을 인식하지 못해서)
반면 yeast와 같이 비교적 짧은 intron을 가지는 생명체는 바로 intron을 인식해서 intron을 제거하게 되는데 이러한 과정을 intron definition에 의한 Splicing이라고 한다. 이 경우에는 intron을 인식하지 못해서 일어나는 intron retension이 가장 흔한 오류이다.
Splice code
그렇다면 어떤 기준으로 alternative splicing을 하게 될까? exon 쇼핑이 아니니 지 좆대로 고르지는 않을 것이다. Splice를 하라고 알려주는 Splicing code를 인식해서 Alternative Splicing을 수행하게 된다. 생명체 내에는 pseudoexon도 존재하는데 이러한 exon은 Splice code를 가지고 있지 않아서 Splice되지 않는다.
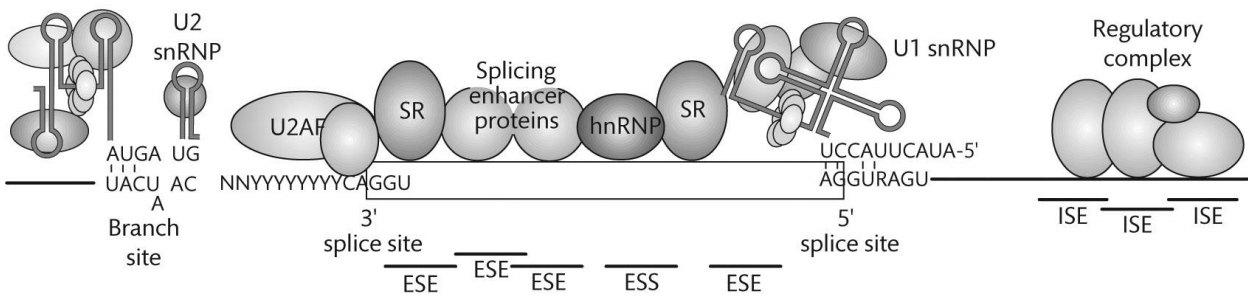
Splice code에는 크게 Splicing Enhancer와 Splicing Silencer가 존재하며 이들은 각각 존재하는 위치에 따라 exonic과 intronic으로 구분된다.
Splicing enhancer에 SR protein이 SE에 결합하면 Spliceosome을 안정화 시켜 Splicing을 activation시킨다. (5'ss의 U1snRNP나 3'ss의 U2AF를 안정화시키는 등...)
Splicing silencer는 loop를 형성하거나 repressor molecule로 RNA를 코팅하여 exon을 인식하는 것을 방해한다. 대표적으로 hnRNP가 그런 역할을 수행한다.
SR protein이나 hnRNP같은 splicing factor가 너무 많아지면 Splicing이 정상적으로 수행되는데 방해가 될 수 있기 때문에 이들은 autoregulation 기전을 가진다.

| SR protein이 많을 때 | hnRNP가 많을 때 |
| poison exon(premature translation termination codon을 만들어 종결)의 ESE에 SR이 결합해 poison exon이 포함된 mRNA가 생성됨 -> SR protein ↓ | hnRNP의 특정 exon이 silencing되어 splice 되지 않음 -> hnRNP ↓ |